XL-MS (Proteinové zesíťování)
Tato metoda je založena na zesíťování proteinu pomocí činidel o specifické délce, které po jejich vazbě na protein a následné hmotnostně spektrometrické detekci dávají informace o vzdálenostech dvou postranních řetěžců aminokyselin. Touto technikou je možné detekovat a strukturně charakterizovat protein-proteinové interakce, nebo charakterizovat vzdálenosti v rámci struktury individuálního proteinu.
Kontaktní osoba: Petra Junková
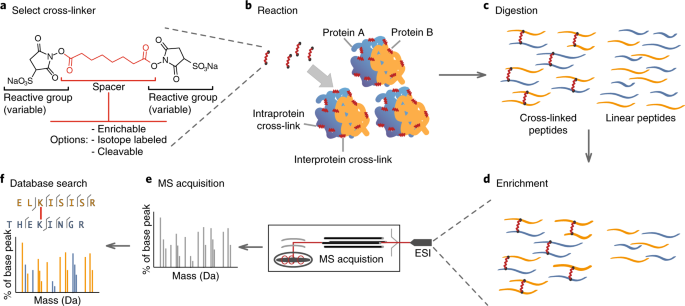
Požadavky:
- Izolované proteiny nebo proteinové komplexy v roztoku
- Sekvence proteinů spolu s informací o jejich modifikacích
- Výběr vhodného zesíťovacího činidla po diskuzi s operátorem
- Vhodný pufr pro provedení zesíťovací reakce záleží na použitém zesíťovacím činidle; činidla obsahující reaktivní skupinu NHS (DSBU, DSPU, DSSBU, DSS, BS3, DSSO) reagují s primárními aminy, proto lze použít pouze pufry, které je neobsahují
- Přesné složení vzorku (koncentrace proteinů, složení pufru, možné kontaminace)
Výstupy:
- List unikátních spojů mezi aminokyselinovými zbytky zkoumaných proteinů ve formátu xlsx či csv
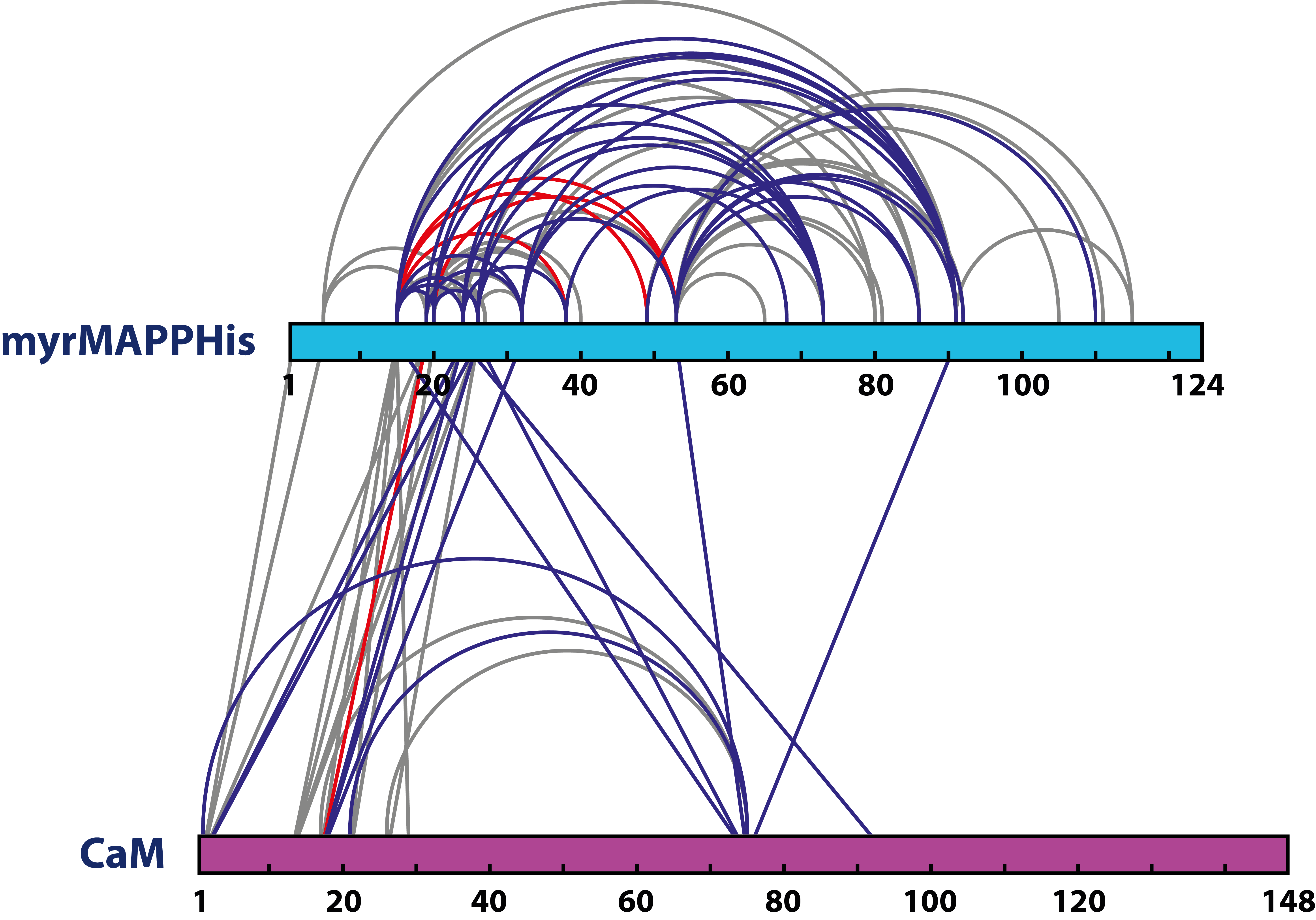
- Schématické znázornění spojů v proteinech získané pomocí xiView ve vektorovém, nebo bitmapovém formátu
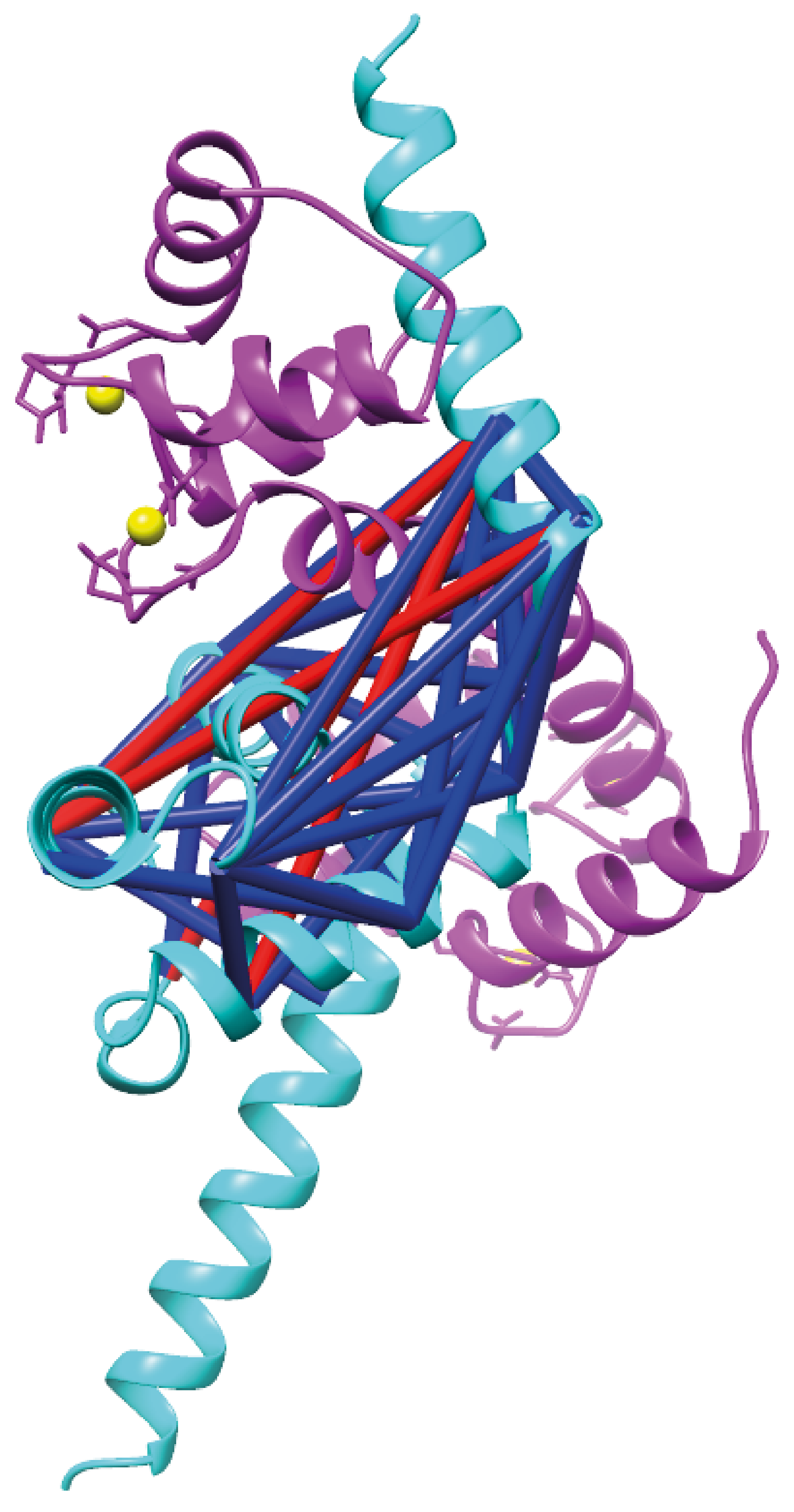
- Validace a zobrazení detekovaných spojů v proteinových strukturách pomocí nástroje Xlink Analyzer (UCSF Chimera) v bitmapovém formátu spolu s analýzou, do jaké míry odpovídají detekované spoje danému strukturnímu modelu proteinu
Očekávaná dodací lhůta:
- 2 – 3 týdny
Pokud máte zájem o XL-MS experiment, prosím kontaktuje nás!

